STD法を用いたタンパク質と低分子の相互作用解析
NM210013
Spin diffusion and Saturation transfer
タンパク質の1H信号を選択的に励起し磁化を飽和させると、飽和された状態がスピン拡散 (spin diffusion) によってタンパク質分子全体に伝わります。さらに、タンパク質と相互作用する低分子のリガンドが溶液中に存在する場合、飽和移動 (saturation transfer) により、リガンドの1H磁化も飽和します (Fig. 1)。
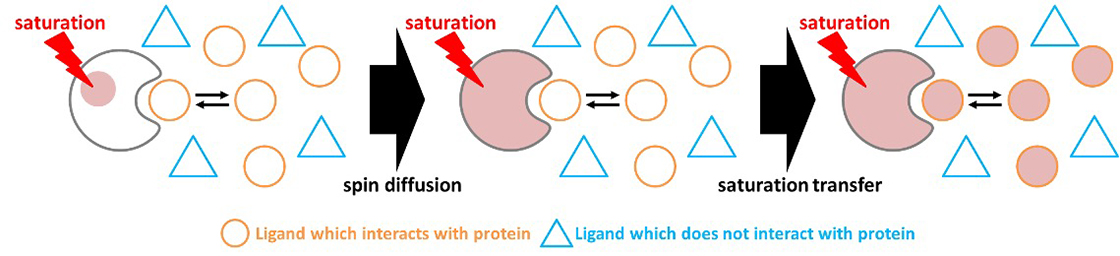
Fig. 1 spin diffusionとsaturation transferの概念図を示す。
タンパク質由来のシグナルを飽和するとタンパク質分子全体へと飽和された状態が広がり、さらにはタンパク質と相互作用する低分子リガンドの磁化も飽和する。なお相互作用しない低分子は飽和しない。
STD (Saturation Transfer Difference)
上記のようにタンパク質を選択励起により飽和させ、さらにそこへリガンドへの飽和移動が起こった場合、タンパク質およびタンパク質と相互作用するリガンドの信号強度は減少します。そこで、選択励起するとき (on resonance) と選択励起しないとき (off resonance) のスペクトルの差をとれば、相互作用しない化合物の信号はキャンセルされ、タンパク質由来の信号およびタンパク質と相互作用するリガンド由来の信号のみが得られることになります。さらに、acquisitionの前にT1ρfilterを加えることで、解析には余分なタンパク質由来の信号を消去することが可能です。※実際のシーケンスでは、on_resonanceとoff_resonanceのスペクトルを交互に取得しています (Fig. 2)。
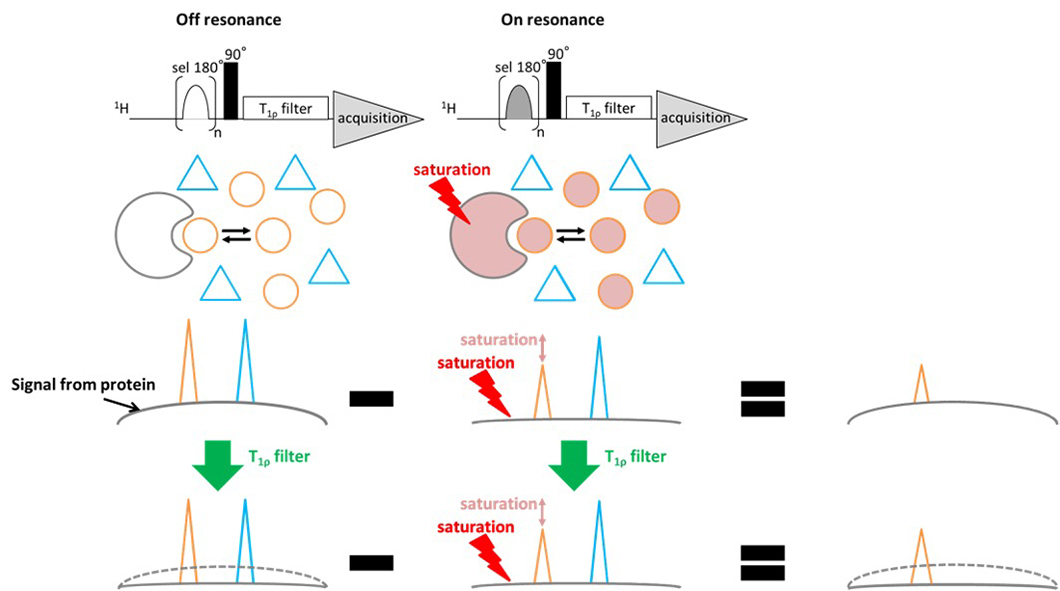
Fig. 2 STD実験のパルスシーケンス及び概念図を示す。
タンパク質をsaturationさせた時とさせない時の差スペクトルを取ることにより、タンパク質とタンパク質と相互作用するリガンド由来のシグナルのみが得られる。またT1ρフィルタにより、タンパク質のブロードなシグナルも低減され、タンパク質と相互作用するリガンドのみのシグナルが得られる。
適用例
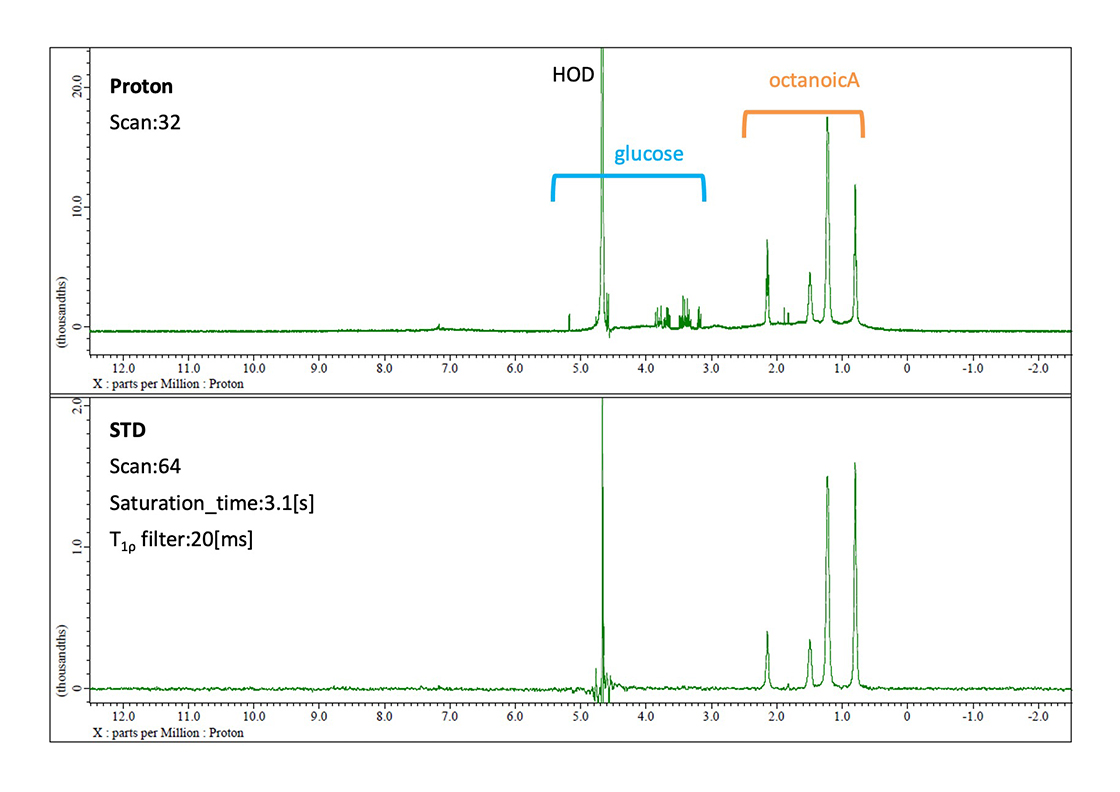
以下に、20 μM HSA (Human Serum Albumin) + 1.0 mM glucose + 1.0 mM octanoic acidの混合溶液について、STD法を用いて相互作用の解析を行った結果を示します (Fig.3)。HSAとの結合活性を持つoctanoic acid由来のシグナルが検出されており、glucose由来のシグナルは検出されていません。このように、STD法はタンパク質などの生体高分子と低分子リガンドとの相互作用を解析するのに有用であり、医薬品のスクリーニングなどに用いられています。
測定条件
| Spectrometer | JNM-ECZL400S |
|---|---|
| Probe | Royal HFX probe |
| Software | Delta v.6.1.0 |
| Temprature | 30dC |
| Pulse sequence | proton.jxp sat_transfer_difference.jxp |
| Sample | 20 μM HSA +1.0 mM glucose +1.0 mM octanoic acid |
Fig. 3 20 μM HSA + 1.0 mM glucose + 1.0 mM octanoic acidのサンプルの、protonスペクトルおよびSTDスペクトルを示す。
プロトンスペクトルではglucoseとoctanoic acid由来のシグナルが両方観測されているが、STDスペクトルではHSAとの結合活性を持つoctanoic acidのみのシグナルが観測されている。
パラメーター設定について
STDスペクトルを取得する際のパラメータ設定について、注意するべきポイントを以下にまとめます。
・選択励起周波数 (std_sel_onreso) と非照射周波数 (std_sel_offreso)
タンパク質の選択励起周波数 (std_sel_onreso) は、リガンドを含む低分子化合物由来のプロトン信号のない領域を選択する必要があります。通常は0.5[ppm-0.6[ppm]付近のブロードなメチルプロトン領域を励起しますが、その領域にリガンドのシグナルが重なる場合でかつタンパク質のアミドプロトンの信号領域である6-9[ppm]にリガンドの信号が存在しない場合には、この領域を照射することも可能です (Fig. 4)。
非照射周波数 (std_sel_offreso) は、タンパク質の信号のある領域から十分に離れた領域 (例えば-10[ppm]) に設定します。しかし、選択励起周波数 (std_sel_onreso) と離れすぎていると、スペクトルの差を取る際にアーティファクトが発生することがあるため、タンパク質の信号のない領域で可能な限り低磁場な位置 (おおよそ-2[ppm]) に設定した方が良い場合もあります (Fig. 4)。
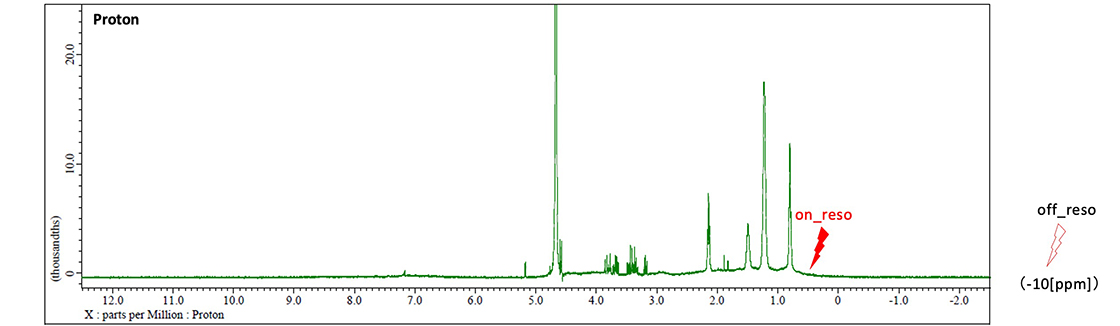
Fig. 4 STD実験における、std_sel_onreso及び std_sel_offresoの化学シフトを示す。
std_sel_onresoはタンパク質のメチル基領域、 std_sel_offresoはタンパク由来のシグナルが存在する領域から離れた領域を設定する。
・飽和時間 (saturation_time)
タンパク質を飽和させる際には、選択励起パルスを連続的に照射します。パルスを照射する時間がsaturation_timeであり、選択励起パルスのパルス幅std_sel_180とstd_loopの積で計算されます。STDの信号を感度良く検出するためには、十分に系が飽和するようなsaturation_timeを設定する必要があり、測定の際にはstd_loopを調整します。Fig.5にHSA + glucose + octanoic acidのサンプルにて、std_loopを変えながら測定した各スペクトルのoctanoic acidの信号の領域 (2.6[ppm]-0.5[ppm]) のアレイを示します。このサンプルでは、std_loop=120程度 (saturation_time=2.5[s]程度) で信号強度が最大となりそれ以上は変化していないことから、std_loopは120以上で設定すればよいということになります。

Fig. 5 std_loopを変化させながらSTDを測定した際のoctanoic acid由来のシグナルが存在するスペクトル領域のアレイを得示す。
std_loop=120程度 (saturation time=2.5 [ms]程度) においてシグナル強度が最大に達している。
・T1ρ filter
タンパク質のブロードなシグナルを消去する目的で、信号を取り込む直前にT1ρフィルタを適用します。T1ρフィルタとは、スピンロックパルスを加えて、横緩和時間 (T2) の短いタンパク質の磁化成分を減衰させる手法です (ここではT2とT1ρは同じ意味と考えて問題ありません)。Fig. 6にHSA + glucose + octanoic acidのサンプルにて、T1ρフィルタの時間を変えながら測定を行った各スペクトルのoctanoic acidの信号の領域 (2.6[ppm]-0.5[ppm]) のアレイを示します。T1ρフィルタによって、タンパク質のブロードな信号が低減されていることが分かります。このサンプルでは、20[ms]程度でタンパク質の信号はほぼ消えており、それ以上長くしたときはリガンドの信号の感度も次第に下がってしまっています。STDのシグナルの感度を保ちながらタンパク質のシグナルを消去するために、適切なT1ρフィルタの時間を設定することが重要です。
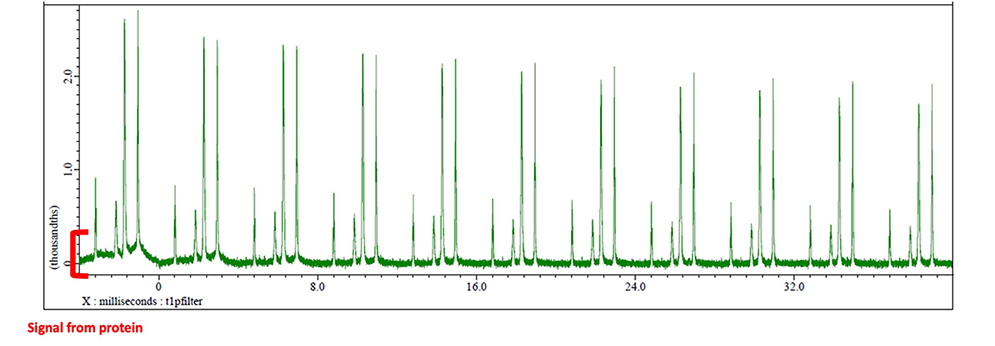
Fig. 6 T1ρ filterの時間を変化させながらSTDを測定した際のoctanoic acid由来のシグナルが存在するスペクトル領域のアレイを示す。
T1ρ filterを長くするほどタンパク質由来のシグナルが消えていき、20[ms]程度でほぼ完全に消えていることが分かる。またそれ以上長くするとSTDのシグナルも次第に低減している。
参考文献
- J. Klein, R.Meinecke, M.Mayer, and B.Meyer, J. Am. Chem. Soc. 1999, 121, 5336-5337.
- M. Mayer and B.Meyer, J.Am.Chem Soc. 2001, 123, 6108-6117.
- J. Yan, A. D. Kline, H. Mo, M. J. Shapiro, and E. R. Zartler, J. of Magnetic Resonance 163, 2003, 270-276.
- このページの印刷用PDFはこちら。
クリックすると別ウィンドウが開きます。 
PDF 837.9 KB
