SiN Window Chipを用いた生物試料の三次元観察
ーSerial Section TEM ー
EM2025-03
はじめに
Serial Section TEMは連続切片内の同一構造を透過電子顕微鏡(TEM)で観察し、その連続断面像を積み重ねて三次元再構成像を作成する手法である。TEMの場合、連続切片の回収にはグリッドが使用されるが、バーによって対象の構造が隠れてしまい(視野カット)、連続観察ができないことがある。そのため、支持膜を張った単孔グリッドに切片回収を行うが、支持膜が破れたり、しわが寄ったりと取り扱いが難しいという課題があった。
日本電子では、高強度の窒化シリコン膜を支持体に用いたSiN Window Chip[1]を開発・販売しており、これを用いることで視野カットのない観察が可能になることから、今回SiN Window Chipを用いてSerial Section TEMを行ったので紹介する。
[1]: Y.Konyuba et al., Microscopy 67 , 367-370 (2018)
Serial Section TEMの流れ
1. SiN Window Chipへの連続切片の回収
ウルトラミクロトームを用いて、厚さ70 nmの連続切片を作製し、SiN Window Chip(図1)に回収した。
試料:ニンジン葉
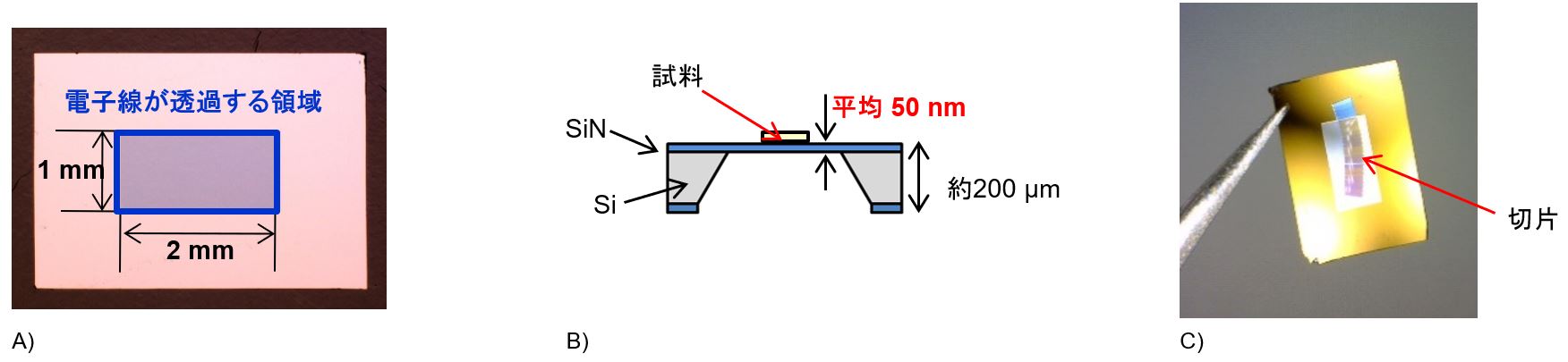
図1 SiN Window Chip
A)外観図 B)断面模式図 C)連続切片を回収したChip
2. 連続切片の撮影
回収した超薄切片からJEM-120i(図2)を用いて観察・撮影を行い、葉肉細胞ひとつ分を網羅する177枚※の連続切片画像を得た。観察にはSiN Window Chip専用の4連試料ホルダーを使用した(図3) 。1度に最大4枚のChipを装填できるので、効率よく観察することができる。また、各Chipの移動はモーター駆動で行われるため、GUI上ですべての操作が完了する。
SiN Window Chip 5枚分

図2 JEM-120i

図3 SiN Window Chip専用4連試料ホルダー
3. 画像処理
TEMで撮影した画像は、Chip上に並んだ連続切片の状態により観察対象(今回は葉肉細胞)の位置や回転が画像ごとにわずかに異なるため、まずScale Invariant Feature Transform (SIFT) を用いた方法[2]で177枚の画像の位置合わせを行った。位置合わせを行うことにより、各画像の視野ずれや切片の回転が補正されている(図4)。
[2] Saalfeld, S. et al., Nat Methods 9, 717–720 (2012)

A)
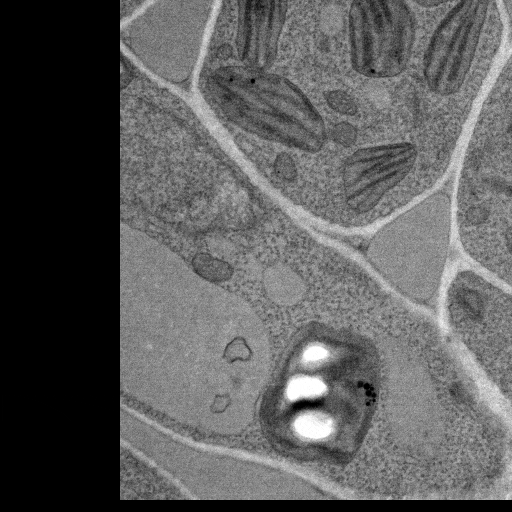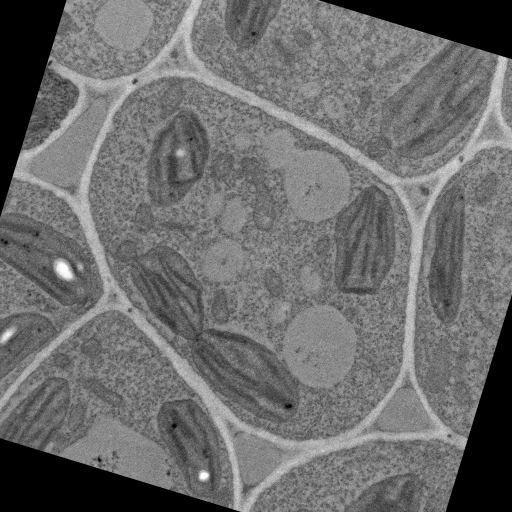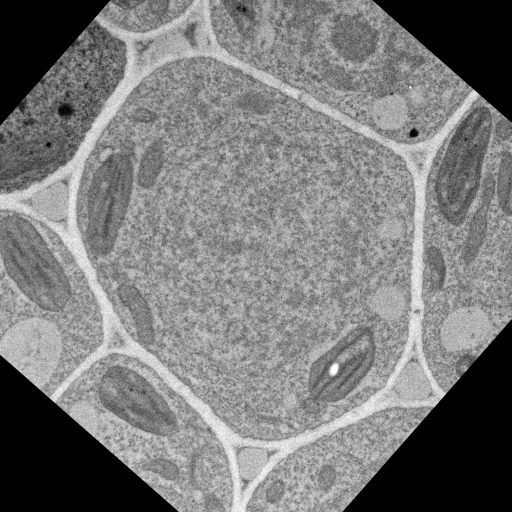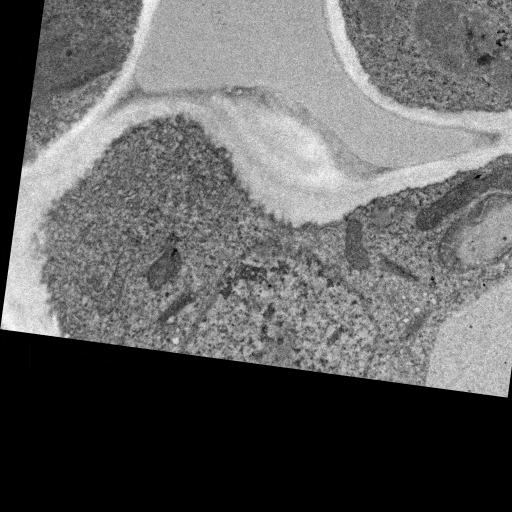
B)
図4 A) SIFTを用いて位置合わせを行った177枚の連続切片画像 B) 位置合わせ後の連続切片画像シリーズ
次に、Deep learning(DL)を用いて連続切片画像から葉緑体と細胞壁のセグメンテーションを行った。DLの教師データとするため、位置合わせ後の連続切片画像から複数枚選択し、手作業にて葉緑体と細胞壁のアノテーションを行った。図5に順番をランダムに選んだ5枚の切片についてのEM画像とアノテーション結果を示す。学習と推論には、U-net[3]を用いた。教師データは、連続した切片画像5枚、順番をランダムに選んだ5枚、20枚および60枚でそれぞれ作成した。
[3] Ronneberger et al., MICCAI 2015, 234–241 (2015).
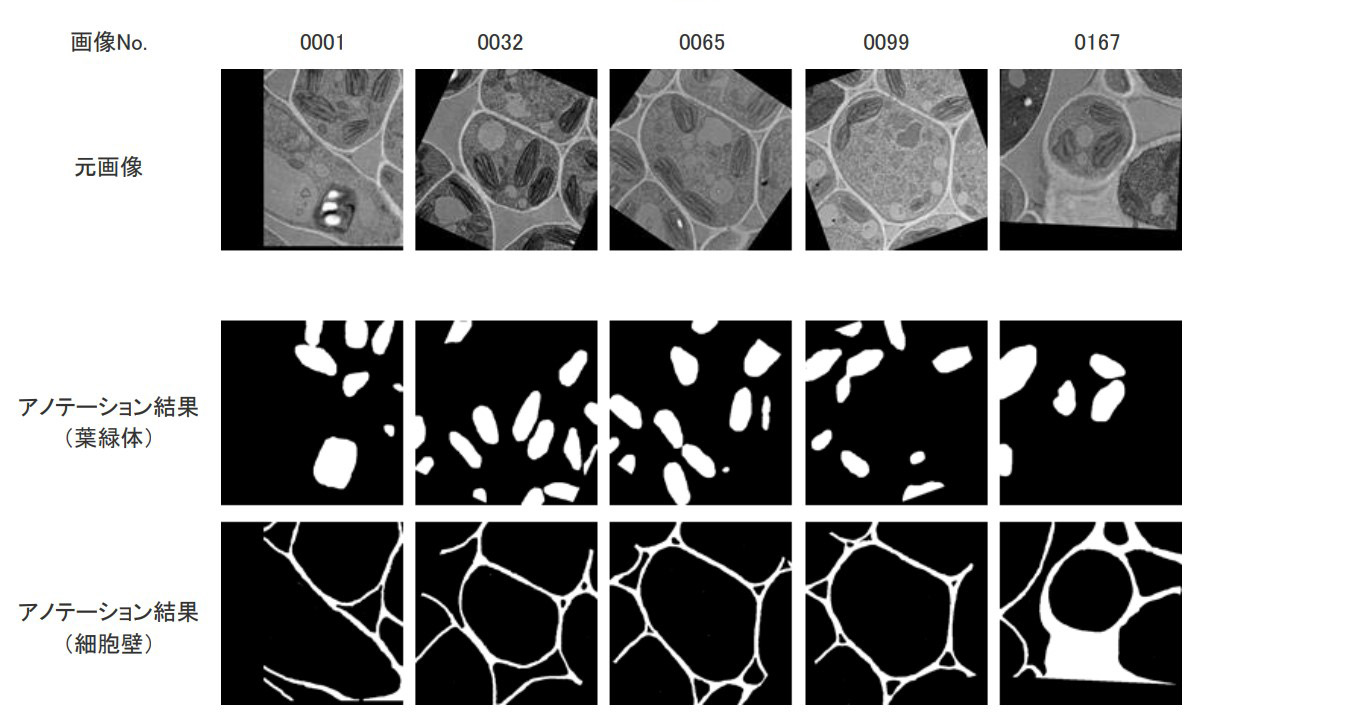
図5 ランダムに選択した画像と手作業によるアノテーション結果
これらの教師データで学習したDLのモデルを用いて葉緑体と細胞壁の推論を行った結果、概ね正しい推論結果を得ることができた(図6)。
葉緑体

細胞壁
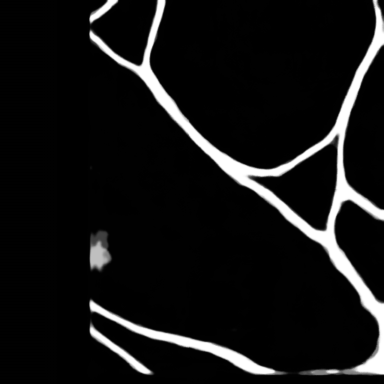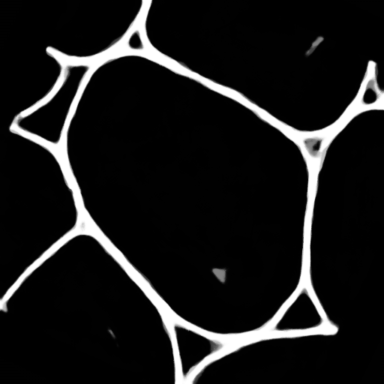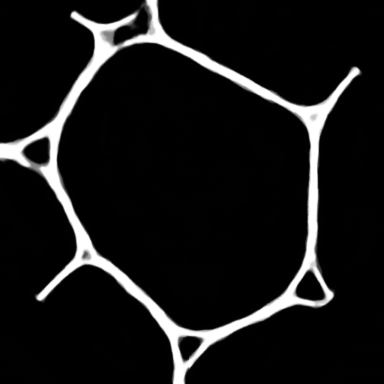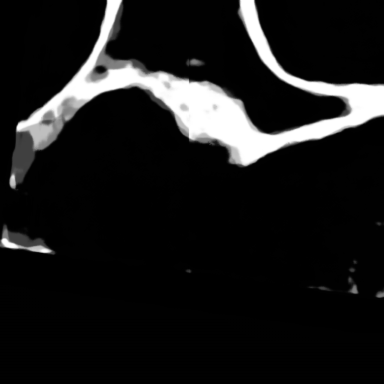
学習:約40分
177枚分の推論:約15分
図6 U-Netによる推論結果
4. 推論精度の評価
セグメンテーションの推論精度を定量的に評価するため、DL分野でよく用いられているF1スコア※を計算した。教師データが5枚の場合、連続した5枚の切片画像よりもランダムに選択した5枚の切片画像を用いて学習した方が推論精度が高いことが分かる(図7、8)。
F1スコア:適合率と再現率の調和平均。1に近いほど推論結果が正しいとされる。

図7 Deep learningによる推論結果精度の比較
ランダムに選択した画像を用いた教師データの場合、画像枚数を増やすことで推論精度が向上するものの、20枚以上ではF1スコアの増加は飽和することが分かった(図8)
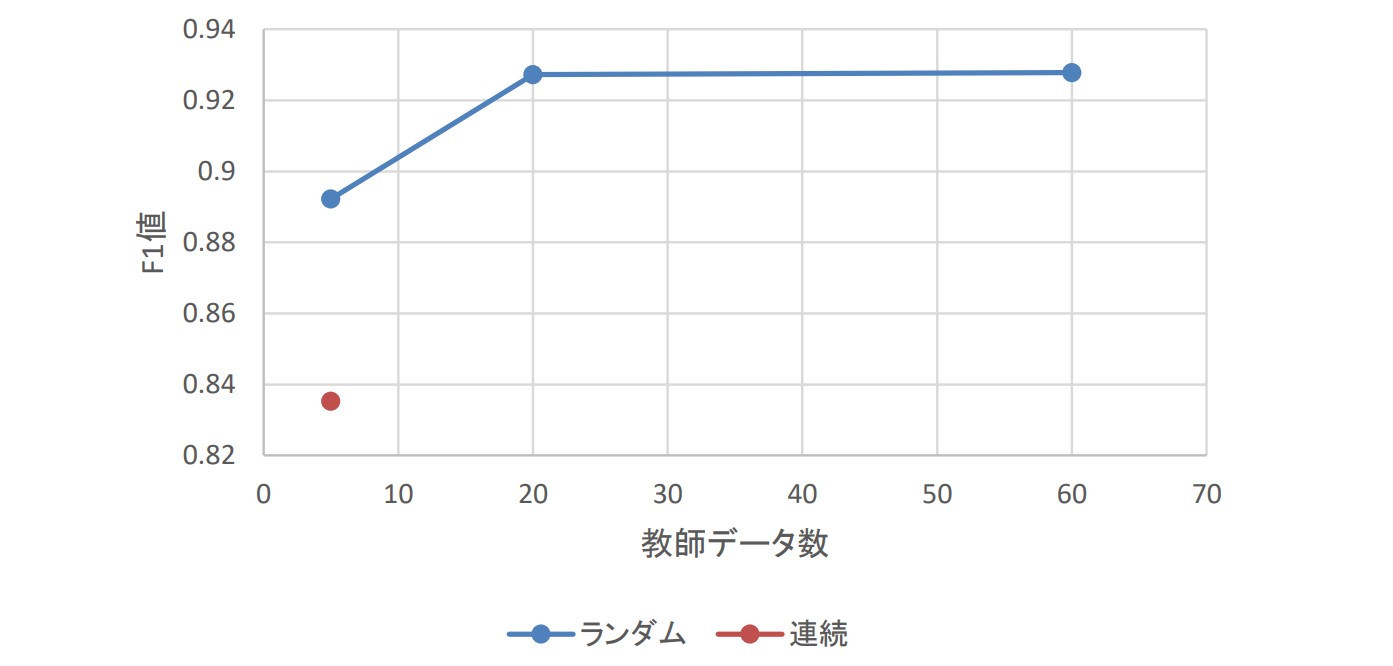
図8 教師データ枚数とF1スコアの関係
5. 三次元再構成
セグメンテーション結果から三次元再構成を行った。三次元再構成像を作ることで、葉肉細胞全体と葉緑体の形状および分布を可視化することができた(図9) 。またDLを用いたことにより、セグメンテーション作業を効率よく行うことができるようになった。
細胞体体積: 約288 µm3
葉緑体積:約63 µm3
葉緑体の割合: 約22%
再構成範囲(x, y, z) :9.4, 9.4, 12.4 µm
ピクセルサイズ: 18.4 nm/pixels
図9 三次元再構成像(動画)
TEMは分解能が高いので、膜構造も明瞭に観察することができる。そこで、より複雑な構造である葉緑体内部のチラコイド膜を対象にセグメンテーションを行い、チラコイドが葉緑体内部全体に広がっている様子が確認できた(図10)。
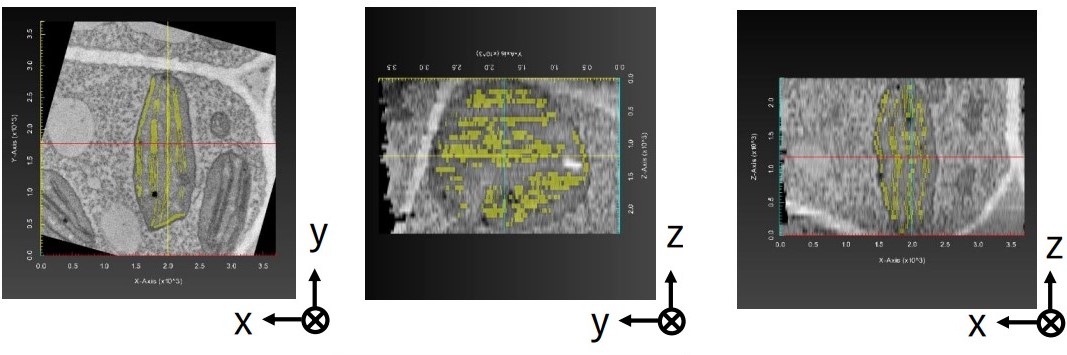
葉緑体体積:約3.19 µm3
再構成範囲(x, y, z) :3.7, 3.7, 25 µm
ピクセルサイズ: 3.6 nm/pixels
図10 葉緑体とチラコイド膜の任意断面像(上段)と三次元再構成像(下段)

