ガスクロマトグラフ-高分解能質量分析および核磁気共鳴による有機合成化合物構造の確認を目的としたmsFineAnalysis AIの活用
- EI/FI測定 -
MSTips : No.463
はじめに
ガスクロマトグラフ-高分解能質量分析 (GC-HRMS) と核磁気共鳴分光分析(NMR)の両方を用いた測定は有機合成化合物の化学組成と化学構造の同定手法として広く利用されている。
GC-HRMSであるJMS-T2000GCは、電子イオン化 (EI) 法およびソフトイオン化の一つである電界イオン化 (FI) 法の両方を使用できるだけでなく、真空を破ることなくEIとFIの切り替えが可能な共用イオン源を有している。この2つのイオン化法による測定結果をmsFineAnalysis AIで解析すると、化学組成以外に化学構造を推測できるため、NMR測定方法の決定およびNMR測定結果の解析に役立つ。そしてGC分離により、サンプルが単成分もしくは混合物から成るかを確認できる。NMR装置であるJNM-ECZ600に搭載可能なROYALPROBE™ HFXはHF側(1H, 19F側)のチューニングを1核、2核それぞれで行うことができ、測定によって使いわけることができる。それによりLF側 (13C等) と合わせて3核の同時チューニングを実現したC, H, Fから成る有機合成化合物に対する強力な化学構造決定ツールである。
本MSTipsでは、JMS-T2000GCとmsFineAnalysis AI、そしてJNM-ECZ600とROYALPROBE™ HFXを用いて有機合成化合物の化学組成と化学構造を解析した事例について報告する。
測定
市販のFluoxetineをサンプルとした。1 mg/mLメタノール溶液に調製したサンプルをMS測定に用いた。また、10 mg/0.6 mL 重水素化DMSO溶液に調製したサンプルをNMR測定に用いた。GC-HRMSおよびNMRの測定条件の詳細はTable 1に示す。
Table 1. Measurement and analysis conditions
| GC-HRMS | JMS-T2000GC (JEOL Ltd.) |
|---|---|
| GC inlet mode | Split 50:1 |
| GC inlet temperature | 280°C |
| GC Column | ZB-5MS, 30m x 0.25mm, 0.25μm (Phenomenex Inc.) |
| GC Oven | 50°C (1 min) →20°C/min →300°C (5 min) |
| Carrier gas | He, 1.0 mL/min |
| MS Ionization | EI+: 70 eV, 300 μA |
| FI+: -10 kV, 40 mA | |
| MS Monitor ion range | m/z 10-800 |
| Analysis software | msFineAnalysis AI (JEOL Ltd.) |
| NMR | JNM-ECZ 600 (JEOL Ltd.) |
|---|---|
| Proton observed frequency | 600 MHz |
| Probe | ROYALPROBETM HFX (JEOL Ltd.) |
| Method | 1H, 19F, 13C {1H,19F} , 1H-13C HSQC, 1H-13C HMBC |
| Analysis software | NMR software Delta 5.3.3 (JEOL Ltd.) |
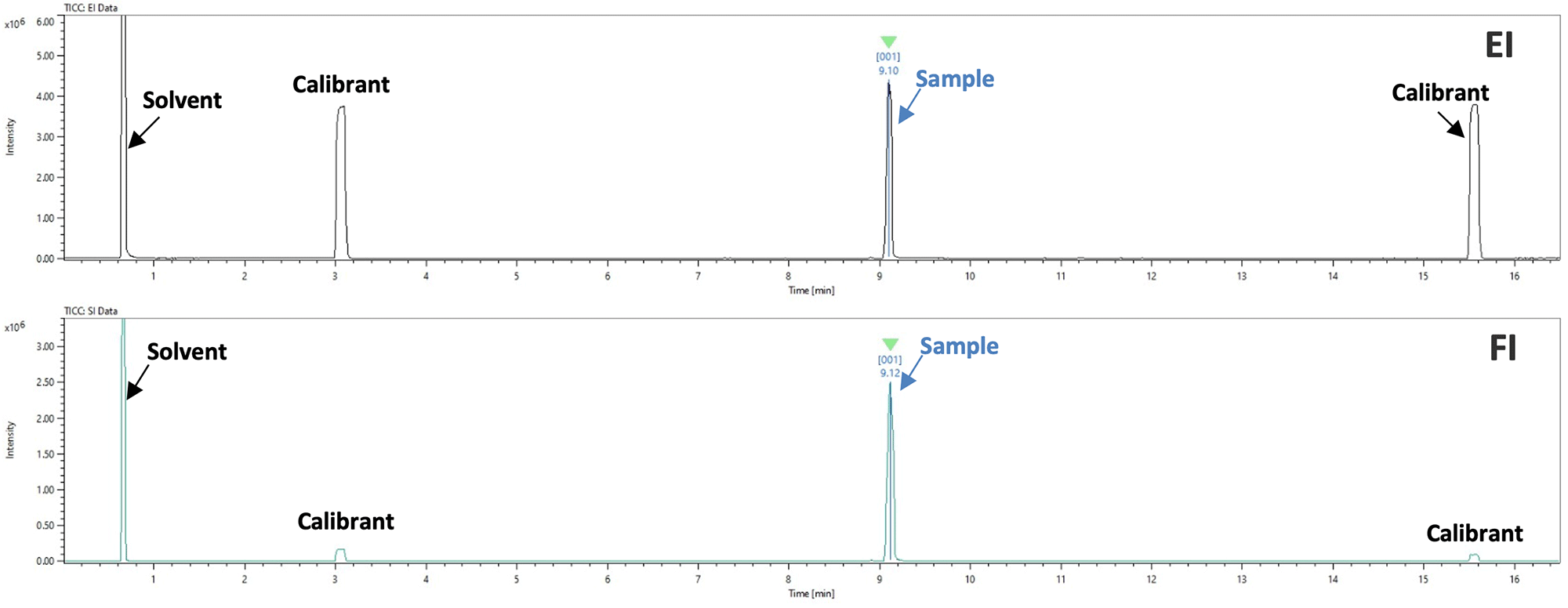
Figure 1. TICCs (top: EI, bottom: FI)
測定結果および考察
EIとFIのトータルイオンカレントクロマトグラム (TICC) をFigure 1に示す。EIとFIのTICCからサンプルピーク1つが同一リテンションタイムで確認された。その他にクロマトグラムピークは検出されなかったため、本サンプルは単一成分から成ることが確認された。msFineAnalysis解析結果をFigure 2に示す。自動解析結果により、Fluoxetineが最有力候補として表示された。FIマススペクトルから確認された分子イオンの化学組成はC17H18F3NOと推定された。分子イオンのアイソトープパターンは、推定されたC17H18F3NOのアイソトープパターンとよく一致した。また、EIフラグメントイオンの化学組成はC17H18F3NOと矛盾のない結果を示した。さらに、NISTデータベース検索でもFluoxetine が一番高い類似度を示した。
次に、NMR結果によるMS結果の確認を行った。msFineAnalysis解析結果を基に考えたNMR測定核種および測定方法によるNMR測定を行った。
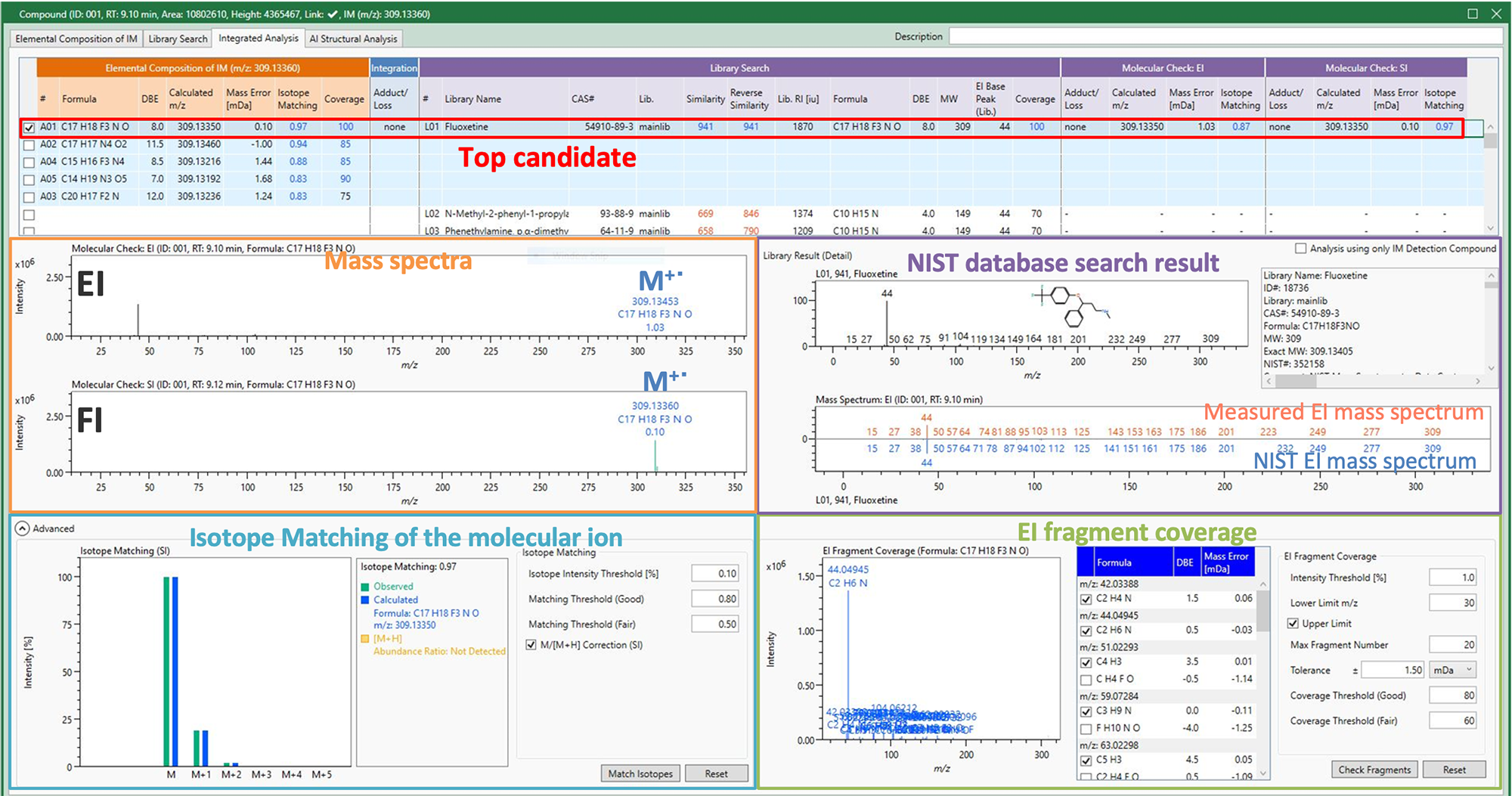
Figure 2. The msFineAnalysis AI results
msFineAnalysis解析結果に基づき19F, 1H, 13C NMR測定を行った。具体的には、一次元19F, 1H, 13C {1H,19F} 及び二次元HSQC (Heteronuclear Single Quantum Coherence) とHMBC (Heteronuclear Multiple Bond Correlation) 測定を実施した。
得られた各NMRスペクトルをFigure 3に示した。図中には参考文献1に示される典型的なfluoxetineの合成経路を基に、重要な帰属を赤字で示した。その他のNMRシグナルも含めて、Figure 4に示すfluoxetineの化学構造を示唆した。
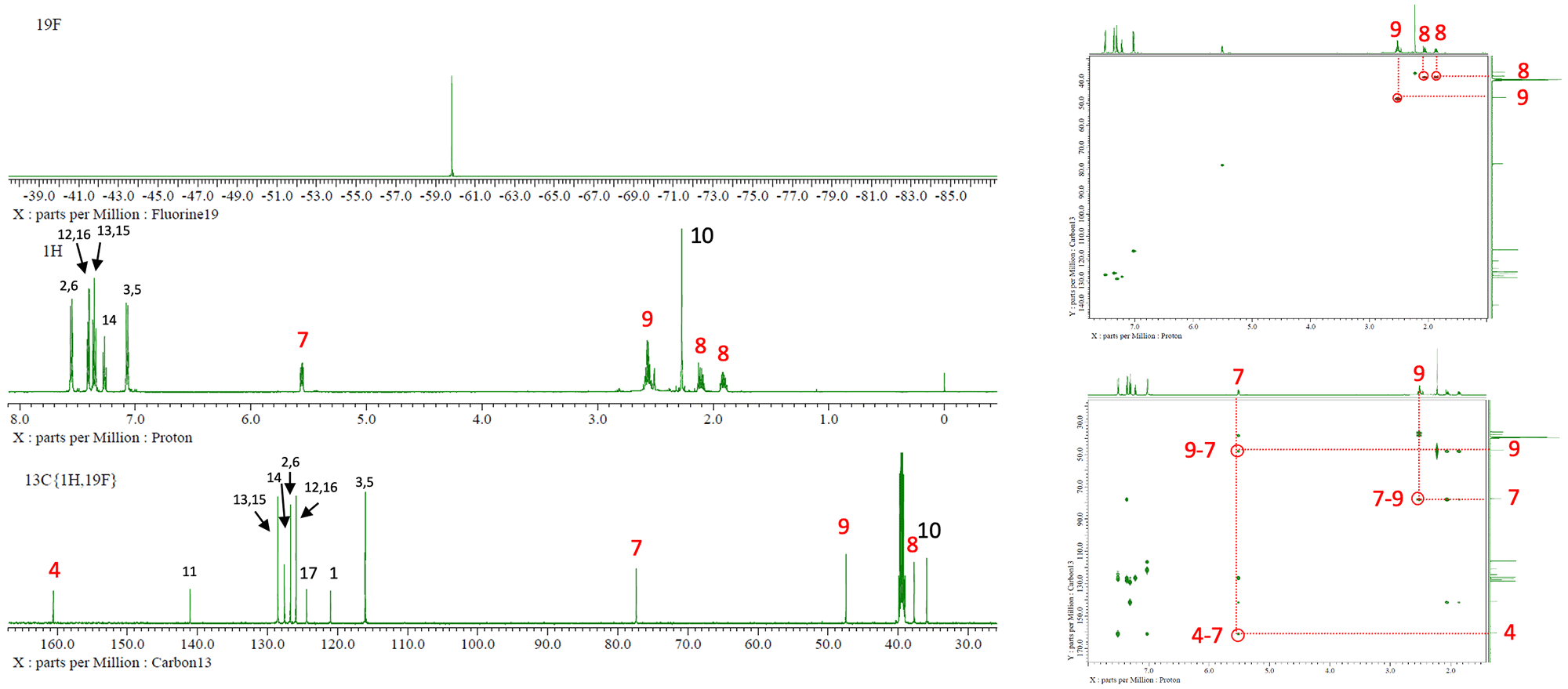
Figure 3. NMR spectra
Left: 19F spectrum (top), 1H spectrum (middle), 13C {1H, 19F} (bottom)
Right: HSQC (top), HMBC (bottom)
| Position | 13C / ppm | 1H / ppm | JF-C/ Hz |
|---|---|---|---|
| 1 | 121.6 | 35 | |
| 2 | 127.3 | 7.55 | 6 |
| 3 | 116.6 | 7.06 | |
| 4 | 161.1 | ||
| 5 | 116.6 | 7.06 | |
| 6 | 127.3 | 7.55 | 6 |
| 7 | 77.9 | 5.55 | |
| 8 | 38.3 | 1.91/2.10 | |
| 9 | 48.1 | 2.56 | 2.27 |
| 10 | 36.6 | ||
| 11 | 141.6 | ||
| 12 | 126.5 | 7.41 | |
| 13 | 129.1 | 7.35 | |
| 14 | 128.1 | 7.27 | |
| 15 | 129.1 | 7.35 | |
| 16 | 126.5 | 7.41 | |
| 17 | 125.0 | 271 |
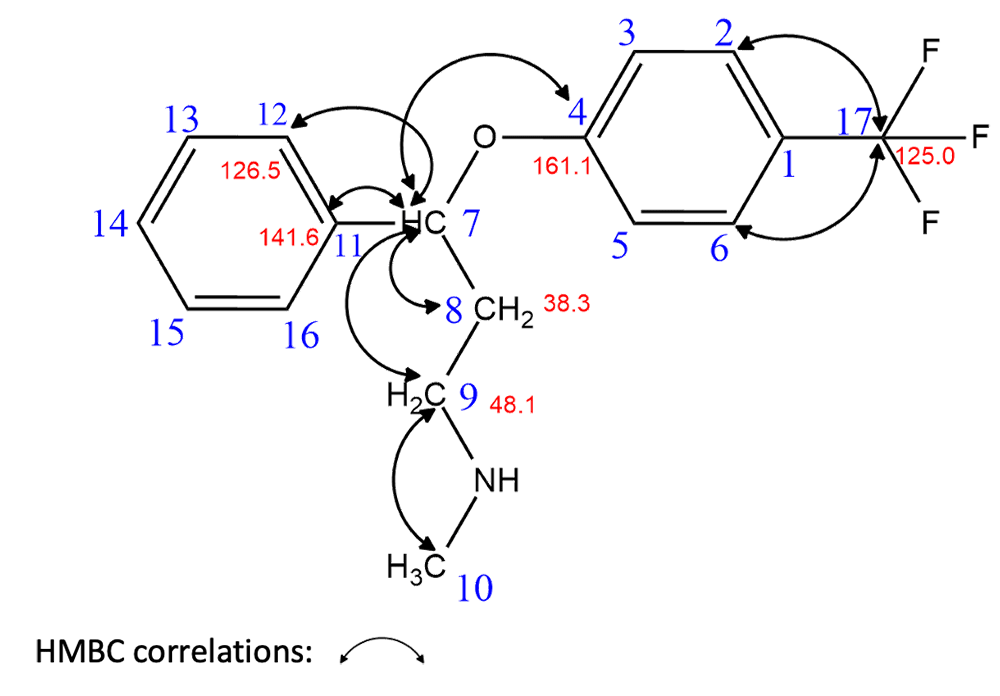
Figure 4. Chemical sift table and HMBC correlations on the chemical structure
まとめ
各測定結果からFigure 5に示す情報を得ることができた。すなわち、EIおよびFIを用いたGC-HRMS結果から、サンプルが単一化合物から成ること、またその化合物の化学組成および推定化学構造を得ることができた。また、GC-HRMS結果から推定された化学組成を基に行ったNMR測定の結果では骨格構造および官能基の位置に関わる情報を得ることができた。今回のサンプルではmsFIneAnalysis AIの解析結果から正しい化学構造であるfluoxetineを推定でき、かつNMR結果により正確に確認された。
本MSTipsで示したJMS-T2000GC + msFineAnalysis AIおよびJNM-ECZ600 + ROYALPROBE™ HFXを用いた有機合成化合物の化学構造の同定方法は、幅広い有機合成化合物のすべての合成段階での利用が期待される。

1. JMS-T2000GC with EI/FI and msFineAnalysis AI
- Confirm the sample is consisted from mixture or unique compound.
- Estimate chemical formula from exact mass of the molecular ion.
- Check the isotope pattern matching and EI fragment ions pattern matching.
- Check the DBE.
- Predict the chemical structure.
2. JNM-ECZ600 with ROYALPROBE™ HFX
- Measure each detectable element of estimated chemical formula.
- Narrow down the predict compounds while assigning NMR spectra.
- Determine the skeletal structure and functional groups.
- Check the enantiomeric purity.
Figure 5. Measurement and analysis procedures of synthetic compounds
参考文献
1) Cody J. Wenthur, Megan R. Bennett, and Craig W. Lindsley., ACS Chem Neurosci. 2014 Jan 15; 5(1): 14–23. doi: 10.1021/cn400186j



