シリアルセクションTEM
シリアルセクションTEM
Serial Section TEM
[目次:理論(電子の散乱/回折/結像)]
樹脂に包埋した試料から連続的に作製した超薄切片を撮影して得られる多数のTEM像を用いて、試料の三次元構造を再構成する手法。細胞や組織の構造を、µm3~mm3の体積かつnmオーダーの解像度で三次元的に可視化することができる。
図1にシリアルセクションTEMの手順を示す。樹脂に包埋した試料から、ウルトラミクロトームで厚さ40~100 nmの超薄切片を連続的に切り出す。切り出した切片をカーボン膜が貼られた単孔グリッドの上に並べ、各切片の同一視野をTEMで順次、観察し撮影する。撮影したTEM像の位置合わせをしてから、目的の構造 (器官) 部分のみを抽出 (セグメンテーションと呼ばれる) し、それらの像を積み重ねることで目的の三次元構造を再構成する。SEMにおけるアレイトモグラフィーと類似の手法である。
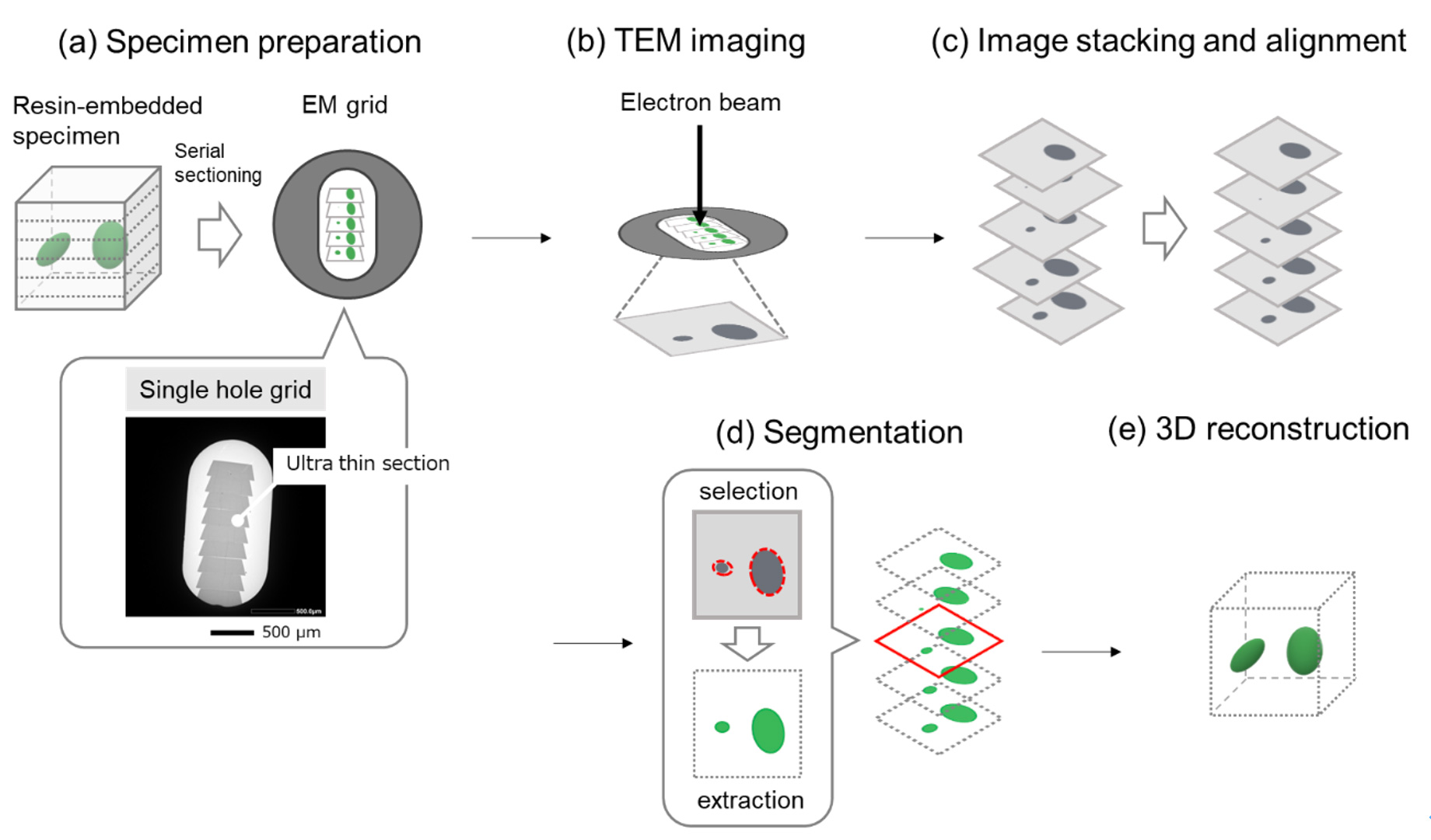
図1 シリアルセクションTEMの手順:
(a) 樹脂に包埋した試料から、ウルトラミクロトームで連続的に切片を切り出し、グリッドの上に並べる。(下部) カーボン膜が貼られた単孔グリッドに連続超薄切片を載せた様子
(b) 各切片の同一視野のTEM像を順次、撮影する。
(c) 撮影したTEM像の位置合わせを行う。
(d) 目的の構造 (器官) のみを抽出 (セグメンテーションと呼ばれる) する。
(e) セグメンテーションした画像を積み重ねることで三次元構造を再構成する。
◆上のボックス内の再生ボタンをクリックするとムービーが始まります。 (30秒) ◆
図2 ニンジンの葉の葉緑体(緑)と 細胞壁(白)の三次元再構成像:
細胞壁の内側にある多数の葉緑体を確認することができる。三次元再構成範囲: (x,y,z) = (9.4, 9.4, 12.4) µm, 試料: ニンジンの葉、装置: JEM-1400Flash、加速電圧: 120 kV、撮影倍率: x4,000、撮影枚数: 177枚
試料支持膜にはカーボン膜以外も使用されることがあり、この場合は窒化ケイ素膜 (SiN Window Chip) を使用した。
Serial Section TEM (transmission electron microscopy) is a reconstruction method of the three-dimensional (3D) structure of a specimen using many TEM images of ultra-thin sections prepared serially from a resin-embedded specimen. The method enables us to visualize 3D structures of cells and tissues of a volume of µm3 to mm3 with a resolution of nanometer order.
Fig. 1 shows the procedure of Serial Section TEM. Ultra-thin sections are serially sliced from a resin-embedded specimen using an ultramicrotome, with a 40 to 100 nm thickness of each section. The prepared sections are lined up on a single hole grid with a carbon film fixed, and the images of the same field-of-view of each section are successively taken by TEM. Then, positions of all the TEM images are correctly adjusted, the target structural part (organ) is extracted (called "segmentation"), and by stacking these images the desired 3D structure is reconstructed. Serial Section TEM is a similar method to Array Tomography in SEM (scanning electron microscopy).

Fig. 1. Procedure of Serial Section TEM
(a) Ultra-thin sections are serially sliced from a resin-embedded specimen using an ultramicrotome, and then the sections are lined up on a single hole grid with a carbon film fixed. Bottom: the aligned ultra-thin sections.
(b) TEM images of the same field-of-view of each section are successively taken.
(c) Positions of all the TEM images are correctly adjusted.
(d) Only the target structural part (organ) is extracted (called "segmentation").
(e) 3D structure of the desired part is reconstructed by stacking these images.
Fig. 2. 3D-reconstructed image of chloroplasts (green) of carrot leaves and cell walls (white).
Many chloroplasts present inside the cell walls are clearly seen.
Size of 3D reconstruction: (x, y, z) = (9.4, 9.4, 12.4) µm, Specimen: carrot leaves, Instrument: JEM-1400Flash, Accelerating voltage: 120 kV, Magnification: ×4000, Number of captured images: 177. For a specimen supporting film, not only a carbon film, but also the other films can be used. In this case, a silicon nitride film (SiN Window Chip) was used.
関連用語から探す
説明に「シリアルセクションTEM」が含まれている用語






